2021年7月16日,植物遗传育种领域的传统期刊Theoretical and Applied Genetics的134卷第8期上发表了我校棉花遗传改良团队题为“Genome-wide association mapping for agronomic traits in an 8-way Upland cotton MAGIC population by SLAF-seq”的论文,该研究利用MAGIC群体和全基因组关联分析解析和挖掘了部分棉花重要农艺性状的遗传位点和基因。
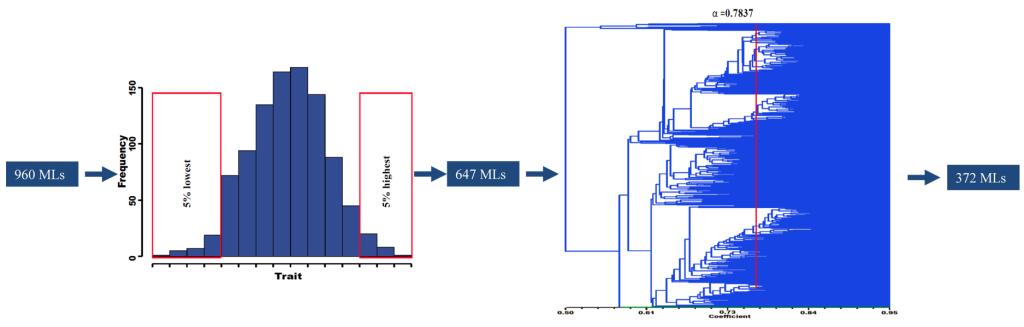
供图1. MAGIC群体筛选过程
该研究基于一个8亲本的陆地棉MAGIC(multi-parent advanced generation intercross)群体(长江大学李定国创建),从960份MAGIC自交系中通过表型和SSR基因型挑选出具有代表性的372份材料(SMLs)。利用SLAF-seq技术对372份材料和8个亲本进行SNP基因型检测,筛选获得60,495个有效SNPs;结合3年6个环境的表型数据,对9个重要农艺性状进行GWAS,以连锁不平衡衰减区间作为参考值,共鉴定到117个QTL位点;有3个QTL位点在多个环境中表现稳定,11个QTL位点与多个性状相关。利用基因组织表达量数据,从QTL候选区间鉴定到154个与关联农艺性状的相关组织特异表达的基因。另外,参考已有的棉花基因功能研究报道,从QTL候选区间内筛选出8个功能已知的候选基因。其中,GhACT1和GhGASL3两个基因已报道与棉花纤维发育相关,位于纤维伸长率的QTL候选区间内。
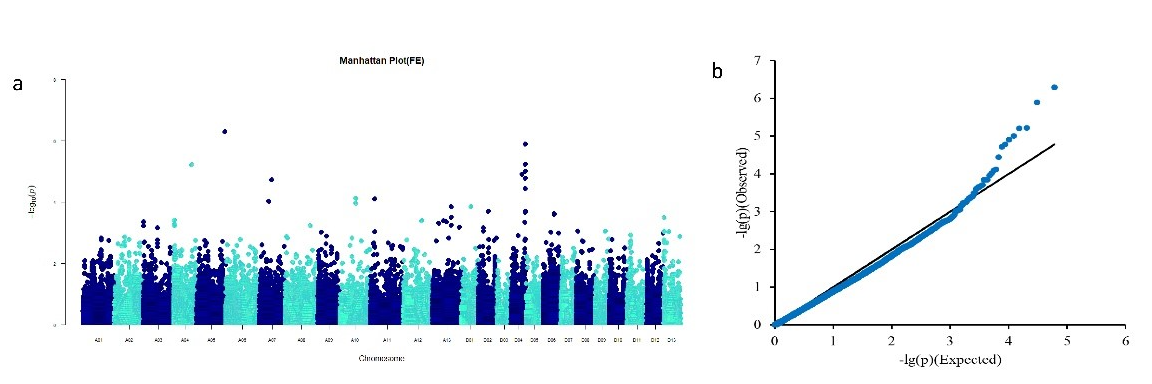
供图2. 纤维伸长率的QTL定位
棉花作为重要的经济作物,关系到纺织业和民生,其农艺性状决定棉花的经济效益,这些性状受复杂的多基因控制,挖掘这些性状的遗传基础对开展棉花遗传育种及品种改良具有重要指导意义。
该论文第一作者为已毕业博士黄聪,现工作于宜昌市农业科学研究院,主要从事特色蔬菜品种选育和栽培技术研究,承担省级项目2项,市级项目1项,编写实用技术书籍2部,获湖北省科技进步二等奖、宜昌市五四青年奖章等荣誉。我校林忠旭教授和长江大学李定国副教授为共同通讯作者。该项工作得到了国家自然科学基金的支持(项目批准号31371674)。
全文链接:https://link.springer.com/article/10.1007/s00122-021-03835-w


